Si es participante o familiar de un participante y aún no hemos contactado con usted, significa que el análisis de su caso continúa en curso.
Debe tener en cuenta que se trata de casos de alta complejidad, tanto desde el punto de vista clínico como genómico, lo que puede requerir más tiempo.
Agradecemos su comprensión.
Se han constituido 5 grupos de trabajo (GdT) para dar apoyo en el adecuado cumplimiento de los objetivos.
Quiénes somos y por qué de este grupo
Tras el análisis genómico es necesaria una transmisión de los resultados de forma clara y apropiada. Se ha constituido un grupo de trabajo encargado de la elaboración de un modelo de informe diagnóstico estandarizado dentro del entorno del programa IMPaCT-GENóMICA para su uso en toda la geografía nacional. Este grupo está compuesto por un conjunto de expertos en medicina genómica personalizada y de precisión.
Funciones
Definición y consenso de los apartados que debe contener el modelo de informe diagnóstico para hacer llegar los resultados de la secuenciación del genoma de manera normalizada y homogénea en todo el territorio nacional a los pacientes reclutados, de una manera inteligible.
Tareas
Recolección y análisis de modelos de informes y guías de recomendación existentes a nivel nacional (CIBERER, RAREGENOMICS, URDCAT, NAGEN, etc.) e internacional (EMQN, ACMG, ESHG, etc.) para su adaptación a IMPaCT-GENóMICA.
Definición de la estructura y contenidos estandarizados del informe.
Redacción del modelo de informe diagnóstico.
Modelo a disposición del entorno IMPaCT-GENóMICA.
Creación de un comité de seguimiento para realizar actualizaciones.
Grupo Core
| Nombre | Centro | Comunidad Autónoma |
|---|---|---|
| Miguel Ángel Moreno | Hospital Ramón y Cajal-CIBERER | Madrid |
| Adriana Lasa | Hospital de Sant Pau-CIBERER | Cataluña |
| Alfredo Santana | Complejo Hospitalario Universitario Materno Insular-CIBERER | Canarias |
| Carmen Ayuso | Hospital Fundación Jiménez Díaz-CIBERER | Madrid |
| Francisco Barros | Fundación Pública Gallega de Medicina Genómica-CIBERER | Galicia |
| José Luis Fernández Luna | Hospital Universitario Marqués de Valdecilla | Cantabria |
| Loida García | Complejo Hospitalario Universitario Materno Insular | Canarias |
| Miguel Fernández Burriel | Hospital de Mérida | Extremadura |
| Pablo Carbonell | Hospital Clínico Universitario Virgen de la Arrixaca | Murcia |
| Pilar Rodríguez | Centro de Diagnóstico de Enfermedades Moleculares CBMSO-CEDEM-UAM-CIBERER | Madrid |
| Susana García | Hospital Universitario Clínico San Cecilio | Andalucía |
| Yolanda Martín | Hospital Ramón y Cajal-CIBERER | Madrid |
Quiénes somos y por qué de este grupo
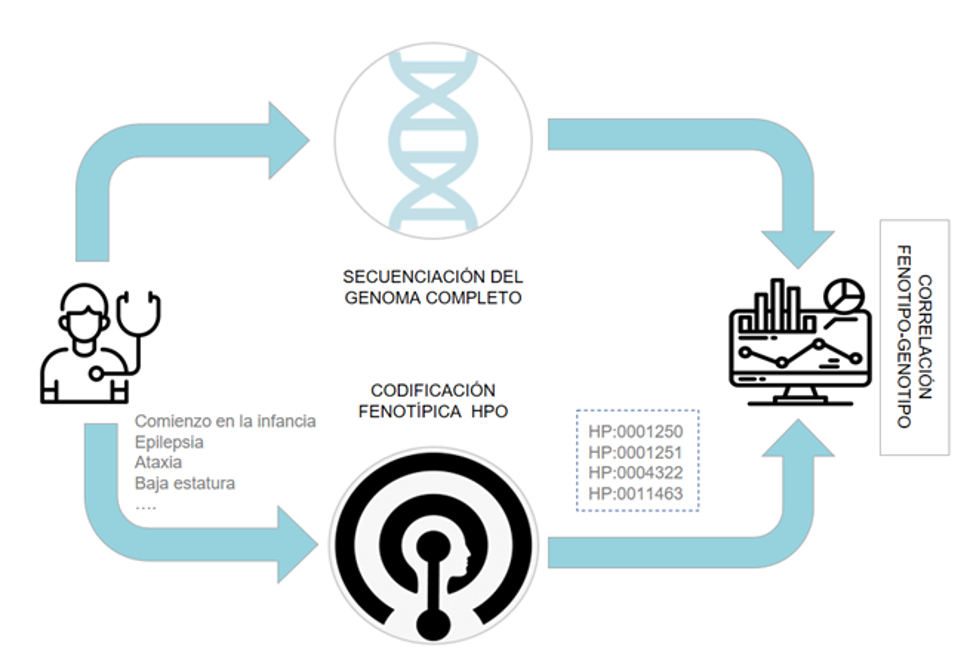
La ontología de fenotipado (Human Phenotype Ontology- HPO) proporciona un vocabulario estandarizado y codificado de anomalías fenotípicas. Esta codificación permite el desarrollo de herramientas informáticas de análisis de variantes que facilitan la correlación fenotipo-genotipo del paciente y es imprescindible para el mejor aprovechamiento de los datos genómicos.
El GdT-Fenotipado HPO centra su trabajo en este aspecto especialmente relevante para la implementación de la medicina de precisión en el ámbito de las enfermedades raras.
Funciones
Difusión de la terminología HPO para aumentar el conocimiento y uso de dicha terminología. Implementación y optimización del fenotipado HPO en la práctica clínica.
Actualmente, está disponible en inglés y se actualiza constantemente. Con el objetivo de su implementación y uso en el sistema sanitario español sería importante su traducción y validación al castellano.
Tareas
Traducción al castellano de las etiquetas (términos preferidos y sinónimos de todos los conceptos HPO).
Traducción de los sinónimos.
Publicación de dichas traducciones.
Establecimiento de un circuito de mantenimiento de las traducciones de las distintas versiones de HPO que se vayan publicando.
Elaboración de documentación (cursos, vídeos, documentos) que incluya los usos actuales, potencialidades y el mecanismo para sugerir cambios en la terminología HPO.
Pilotaje de la implementación e integración de la terminología HPO en un contexto hospitalario.
Testeo de alguna solución de procesamiento del lenguaje natural (PLN) para extracción automática de terminología HPO a partir de la historia clínica.
Grupo Core
| Nombre | Centro | Comunidad Autónoma |
|---|---|---|
| Jaime Cruz | Hospital 12 de Octubre | Madrid |
| Guiomar Pérez de Nanclares | Instituto de Investigación Sanitaria Biocruces Bizkaia | País Vasco |
| Andrea Sariego Jamargo | Hospital Universitario Marqués de Valdecilla | Cantabria |
| Fernando Santos Simarro | Hospital Universitario Son Espases-CIBERER | Baleares |
| Guerau Fernández | Hospital Sant Joan de Déu-CIBERER | Cataluña |
| Raquel Rodríguez | Hospital General de Valencia | Valencia |
Quiénes somos y por qué de este grupo
Existen más de 7000 enfermedades raras y este número va en aumento con el descubrimiento continuo de nuevos genes y nuevas asociaciones fenotipos-genotipos.
El GdT-Comité de expertos tiene como misión crear una Red de expertos en enfermedades raras, que estará formada por profesionales del territorio nacional en todas las áreas de la asistencia de la medicina genómica en este ámbito, tanto a nivel clínico como molecular. Se trata de una Red de expertos y no de instituciones.
Funciones
Creación de una Red de expertos en ER que puedan ser consultados a propósito de casos específicos dentro de IMPaCT-GENóMICA.
Tareas
Establecimiento de los criterios para la creación de la red y la inclusión de expertos.
Identificación y definición de los campos de experiencia: estratificación por campo profesional, grupos de enfermedades, patologías, genes y técnicas diagnósticas.
Establecimiento de los mecanismos de creación, mantenimiento y explotación de la base de datos y de actualización de los contactos de los expertos.
Grupo Core
| Nombre | Centro | Comunidad Autónoma |
|---|---|---|
| Belén Pérez | Centro de Diagnóstico de Enfermedades Moleculares CBMSO-CEDEM-UAM-CIBERER | Madrid |
| Victor Manuel Martínez González | Hospital Universitari Parc Taulí | Cataluñá |
| Aurora Pujol | Instituto de Investigación del Hospital de Bellvitge IDIBELL-CIBERER | Cataluña |
| Eduardo López-Laso | Hospital Universitario Reina Sofía-CIBERER | Andalucía |
| Feliciano Jesús Ramos | Hospital Lozano Blesa-CIBERER | Aragón |
| Guiomar Pérez de Nanclares | Instituto de Investigación Sanitaria Biocruces Bizkaia | País Vasco |
| Miguel Ángel Moreno | Hospital Ramón y Cajal-CIBERER | Madrid |
Quiénes somos y por qué de este grupo
Tras un análisis genómico es muy probable hallar variantes genéticas de las cuales no se conoce su efecto en la salud (variantes de significado incierto) o encontrar incongruencias entre el fenotipo y el genotipo.
El GdT-Validación funcional trabaja en la estructuración de los estudios de genómica funcional de variantes genéticas con el fin de intentar determinar su consecuencia.
Funciones
Identificación de capacidades en las pruebas de validación funcional necesarias para una confirmación diagnóstica.
Tareas
Elaboración de un listado de técnicas experimentales-moleculares, celulares y ómicas junto con aproximaciones in silico disponibles en cada grupo de investigación participante para el análisis de las variantes. Búsqueda de expertos para cubrir este listado de técnicas.
Establecimiento de un modelo de estructura para implementar la genómica funcional según un modelo de diagnóstico traslacional.
Grupo Core
| Nombre | Centro | Comunidad Autónoma |
|---|---|---|
| Francesc Palau Martínez | Hospital Sant Joan de Déu-CIBERER | Cataluña |
| Aurora Pujol | Instituto de Investigación del Hospital de Bellvitge IDIBELL-CIBERER | Cataluña |
| Belén Pérez | Centro de Diagnóstico de Enfermedades Moleculares CBMSO-CEDEM-UAM-CIBERER | Madrid |
| Frederic Tort | Hospital Clínic de Barcelona-CIBERER | Cataluña |
| Javier Corral | Hospital José María Morales Meseguer-CIBERER | Murcia |
| Jordi Surrales | Hospital de Sant Pau-CIBERER | Cataluña |
| Karen Elise Heath | Hospital La Paz-CIBERER | Madrid |
| Matías Morín | Hospital Ramón y Cajal-CIBERER | Madrid |
| Miguel Ángel Martín | Hospital 12 de Octubre-CIBERER | Madrid |
| Víctor Ruíz | Instituto de Investigaciones Biomédicas "Alberto Sols"-CIBERER | Madrid |
Quiénes somos y por qué de este grupo
La compartición de datos genómicos es muy importante para determinar la patogenicidad de las variantes causantes de enfermedades raras (ER). Actualmente existen sistemas que garantizan todos los requerimientos ético-legales para compartir la variabilidad genética imposibilitando la identificación de las personas, lo que permite consultar la frecuencia con la que las variantes se dan en la población e identificar variantes asociadas a la misma enfermedad. Aquí se incluyen bases de datos de almacenamiento genómico como la base de datos European Genome-Phenome Archive (EGA), o los sistemas que permiten la consulta anónima de variantes concretas y los fenotipos que producen (sistemas Beacon).
El GdT-Almacenamiento y data sharing se encarga de facilitar que los datos genómicos secuenciados estén disponibles para la comunidad científica para su uso secundario. La tecnología adquirida para IMPaCT-GENóMICA debería poder ser trasladable a cada grupo de investigación para crear una infraestructura nacional de compartición de datos que impulse la investigación en ER. Trabaja junto con iniciativas similares dentro del CIBERER (Grupo de trabajo en Bioinformática y la plataforma CIBERER-BIER) y con IMPaCT-Data con los que forma un grupo de trabajo mixto. Además, está alineado con el proyecto europeo 1+Million Genomes, en el que España participa y que pretende recopilar la variabilidad genética europea.
Este grupo de trabajo actualmente desarrolla su actividad dentro del contexto de los grupos de trabajo transversales de IMPaCT.

Funciones
Tareas
Encuesta a los coordinadores de las CCAA de enfermedades raras para detectar cuál es la situación actual en cuestión de datos genómicos acumulados y su compartición.
Taller online abierto sobre EGA/Beacon el 27 de enero de 2022, con más de 200 asistentes. La grabación está disponible aquí:https://youtu.be/RI9wCs7JVKw).
Creación de un grupo de trabajo transversal (GdT-T) con el programa IMPaCT-Data para ofrecer soluciones EGA/Beacon acordes a las necesidades de IMPaCT-GENóMICA.
Participantes
| Nombre | Centro | Comunidad Autónoma |
|---|---|---|
| Pablo Mínguez | Instituto de Investigación Fundación Jiménez Díaz-CIBERER | Madrid |
| Ángel Campos | Hospital Universitario la Paz-CIBERER | Madrid |
| Ángela del Pozo | Hospital Universitario la Paz-CIBERER | Madrid |
| Aurora Pujol | Instituto de Investigación del Hospital de Bellvitge IDIBELL-CIBERER | Cataluña |
| Benjamín Rodríguez | Hospital de Sant Pau-CIBERER | Cataluña |
| Carla Escribano García | Hospital de Valladolid | Castilla y León |
| Eliecer Coto | Hospital Universitario Central de Asturias | Asturias |
| Feliciano Jesús Ramos | Hospital Lozano Blesa-CIBERER | Aragón |
| Guerau Fernández | Hospital Sant Joan de Déu-CIBERER | Cataluña |
| Javier Fernández | Hospital 12 de Octubre | Madrid |
| Javier Pérez | Fundación Progreso y Salud-CIBERER | Andalucía |
| Joan Maynou | Hospital Sant Joan de Déu-CIBERER | Cataluña |
| Jorge Amigo | Fundación Pública Gallega de Medicina Genómica-CIBERER | Galicia |
| José Luis Fernández | Fundación Pública Andaluza Progreso y Salud FPS-CIBERER | Andalucía |
| José Luis Villanueva | Hospital Clínic de Barcelona | Cataluña |
| Juan F. Quesada-Espinosa | Hospital 12 de Octubre | Madrid |
| Laura Gort | Hospital Clínic de Barcelona-CIBERER | Cataluña |
| Luis Castaño | Instituto de Investigación Sanitaria Biocruces Vizcaya-CIBERER | País Vasco |
| Luis Pérez | Hospital del Mar-CIBERER | Cataluña |
| Luis Ruiz | Hospital Universitario Marqués de Valdecilla | Cantabria |
| Marc Pybus | Fundación Puigvert | Cataluña |
| María de la Luz Couce | Universidad de Santiago de Compostela-CIBERER | Galicia |
| María Isabel Álvarez | Hospital Clínic de Barcelona-CIBERER | Cataluña |
| Miguel Ángel Moreno | Hospital Ramón y Cajal-CIBERER | Madrid |
| Miguel Fernández | Hospital de Mérida | Extremadura |
| Natalia Castejón | Centro de Diagnóstico de Enfermedades Moleculares CBMSO-CEDEM-UAM | Madrid |
| Raquel Rodríguez | Hospital General de Valencia | Valencia |
| Roberto Zarrabeitia | Hospital de Sierrallana-CIBERER | Cantabria |
| Sergio Fernández | Hospital Ramón y Cajal-CIBERER | Madrid |
| Vicente Ginger | Hospital Clínico Universitario San Juan | Valencia |